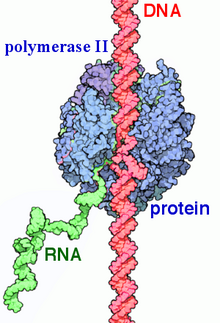
RNAポリメラーゼII
RNAポリメラーゼII(英: RNA polymerase II、略称: RNAP II、Pol II)は複数のタンパク質からなる複合体で、真核生物の細胞核に存在する3つのRNAポリメラーゼのうちの1つである。DNAの転写を触媒し、mRNAの前駆体、snRNAの大部分とmiRNAを合成する。12種類のサブユニットからなる 550 kDaの複合体で、真核生物のRNAポリメラーゼのうちで最もよく研究が行われている。遺伝子の上流のプロモーターに結合して転写を開始するには、ポリメラーゼの他にさまざまな転写因子が必要である。
発見

初期の研究からは、少なくとも2種類のRNAポリメラーゼが存在することが示唆されており、1つは核小体でrRNAを合成し、もう1つは核質で他のRNAを合成すると考えられていた。1969年、ロバート・ローダーとウィリアム・J・ラターはある種のRNAの転写を担う、さらに別のRNAポリメラーゼが核質に存在することを決定的に示した。このことはDEAE-Sepahdexによるイオン交換クロマトグラフィーによって発見され、この技術によってRNAポリメラーゼはI、II、IIIという3つの画分へと分離された。それぞれクロマトグラフィーカラムからの溶出の順序に従って、RNAポリメラーゼI、II、IIIと名付けられた。この発見によって核質に複数の酵素が存在することが示され、RNAポリメラーゼIIとIIIが区別されることとなった。
サブユニット
真核生物のRNAポリメラーゼIIのコア酵素は転写アッセイによってはじめて精製された。精製されたコア酵素は10から12種類のサブユニット(ヒトと酵母では12種類)を含んでおり、特定のプロモーターを認識する能力は持たない。多くのサブユニット間相互作用が知られている。
- RPB1(POLR2A)– ヒトではPOLR2A遺伝子、酵母ではRPO21遺伝子にコードされている。RPB1はRNAポリメラーゼIIの最大のサブユニットである。ポリメラーゼ活性に必須な、最大52個のヘプタペプチドリピート配列(YSPTSPSの7アミノ酸からなるリピート配列)から構成されるC末端ドメイン(CTD)を含んでいる。CTDはトロント大学のC. J. Inglesの研究室とジョンズ・ホプキンズ大学のJ. L. Cordenによって最初に発見された。RPB1は他のいくつかのサブユニットとともにポリメラーゼのDNA結合ドメインを形成し、DNAの鋳型がRNAへ転写される溝を形成する。RPB8と強く相互作用する。
- RPB2(POLR2B)– 2番目に大きなサブユニットであり、少なくとも2つの他のサブユニットとともにポリメラーゼ内部の構造を形成し、酵素の活性部位でDNA鋳型と新生RNAとの接触を維持する。
- RPB3(POLR2C)– 3番目に大きなサブユニット。他のサブユニットRPB11とのヘテロ二量体として存在し、組み立ての際のコアとなる。RPB3はRPB1–5、7、10–12と強く相互作用する。
- RPB4(POLR2D)– POLR2D遺伝子にコードされる4番目に大きなサブユニットで、ストレスからの保護機能を持つ可能性がある。
- RPB5(POLR2E)– ヒトではPOLR2E遺伝子にコードされる。RNAポリメラーゼIIにはこのサブユニットが2分子存在している。RPB5はRPB1、3、6と強く相互作用する。
- RPB6(POLR2F)– 少なくとも2つの他のサブユニットとともに、DNA鋳型を転写しているポリメラーゼを安定化する構造を形成する。
- RPB7(POLR2G)– POLR2G遺伝子にコードされ、転写開始や伸長中のポリメラーゼの安定化する機能を持つと考えられている。RPB7はRPB1、5と強く相互作用する。
- RPB8(POLR2H)– RPB1–3、5、7と相互作用する。
- RPB9(POLR2I)– DNAの鋳型がRNAへ転写される溝はRPB9とRPB1から構成されている。
- RPB10(POLR2L)– POLR2L遺伝子の産物である。RPB1–3、5と相互作用し、中でもRPB3と強く相互作用する。
- RPB11 – ヒトではRBP11サブユニットは3種類存在する。POLR2J (RPB11-a)、POLR2J2 (RPB11-b)、そしてPOLR2J3 (RPB11-c) である。
- RPB12(POLR2K)– RPB3と相互作用する。
組み立て
RPB3はRNAポリメラーゼIIの組み立てに関与している。RPB2とRPB3のサブコンプレックスはサブユニットの合成の直後に出現する。この複合体はその後RPB1と相互作用する。RPB1が複合体に取り込まれると、RPB5やRPB7などの他のサブユニットが取り込まれる。RPB5はRPB6とRPB8と結合し、RPB3はRPB10、RPB11、RPB12を取り込む。RPB4とRPB9は複合体の大部分が組み立てられてから取り込まれる。RPB4はRPB7と複合体を形成する。
α-アマニチンによる阻害
RNAポリメラーゼIIはα-アマニチンや他のアマトキシンによって阻害される。α-アマニチンは多くのキノコに存在する、極めて毒性の高い物質である。キノコ毒は各RNAポリメラーゼに異なる影響を与える。RNAP Iはこの物質に全く反応せず通常に機能し、RNAP IIIは軽度の感受性を示す。しかし、RNAP IIはこの毒素によって完全に阻害される。α-アマニチンはRPB1サブユニットの"funnel"、"cleft"、"bridge α-helix"と呼ばれる領域と強く相互作用し、阻害を行う。
ホロ酵素
RNAポリメラーゼIIホロ酵素とは、生細胞内でタンパク質をコードする遺伝子のプロモーターに呼び寄せられた、RNAポリメラーゼIIの一形態である。RNAポリメラーゼIIと一群の基本転写因子、そしてSRBタンパク質と呼ばれる調節因子から構成される。
組み立てられたホロ酵素の一部は、転写開始前複合体と呼ばれる。それは、この組み立てが転写開始の前に遺伝子のプロモーター領域で起こるためである。メディエーター複合体は、RNAポリメラーゼIIと転写因子の間のブリッジとして機能する。
転写の機構
RNAポリメラーゼのCTD
RPB1のC末端領域はC末端ドメイン(CTD)を形成する。RNAポリメラーゼIIのCTDは、一般的にはTyr-Ser-Pro-Thr-Ser-Pro-Serのリピート配列からなり、最大52個のリピート配列から構成される。CTDはRNAポリメラーゼIIのコアからRNAの出口となるexit channelへと伸び、RNAのプロセシング装置の構成要素と直接的・間接的な相互作用を介してRNAのプロセシング反応を誘導するために効率的な配置となっている。CTDはRNAポリメラーゼIやRNAポリメラーゼIIIには存在しない。CTDはトロント大学のC. J. Inglesの研究室とジョンズ・ホプキンズ大学のJ. Cordenの研究室において、それぞれ酵母とマウスのRPB1サブユニットをコードするDNAのシーケンシングの過程で発見された。CTDには他のタンパク質がしばしば結合し、ポリメラーゼの活性を活性化する。このドメインは、転写開始、RNA転写産物への5'キャップの付加、RNAスプライシングのためのスプライソソームの結合などに関与している。
CTDドメインのリン酸化
RNAポリメラーゼIIは非リン酸化状態(IIA)とリン酸化状態(IIO)の2状態で存在する。2状態間の転換によって、転写の異なる機能が促進される。CTDのリン酸化は6つの基本転写因子のうち1つ、TFIIHによって触媒される。TFIIHの機能の1つは転写開始部位のDNAの巻き戻しであり、もう1つはリン酸化である。転写開始前複合体に加わるのはIIA型のRNAポリメラーゼIIであり、このことは基本転写因子TFIIDのサブユニットTBP(TATA結合タンパク質)に対し、IIA型はIIO型よりも高い親和性で結合することから示唆される。IIO型はRNA鎖の伸長を促進する。伸長の開始はTFIIHを介したリピート配列の5番目のセリン残基(Ser5)のリン酸化によって行われる。新たにリン酸化されたSer5は新生RNA鎖の5'末端へキャップを付加する酵素や3'末端のポリ(A)化部位のプロセシングを行う因子をリクルートする。リピート配列の2番目のセリン残基(Ser2)がリン酸化されると、伸長反応が活性化される。伸長を終結させるためには、脱リン酸化が起こる必要がある。CTDが完全に脱リン酸化されると、RNAポリメラーゼIIは「リサイクル」され、他の転写開始部位で同様の過程を触媒する。
転写と共役した組換え修復
DNAの酸化損傷はRNAポリメラーゼIIによる転写の障壁となり、DNA鎖の切断を引き起こす可能性がある。RNAを鋳型とした、転写と関係した組換え過程は、DNA損傷からの保護を行う可能性がある。細胞周期のG1/G0期の間、細胞は活発な転写領域内部の二本鎖切断部位へ相同組換え因子を集合させる。この過程は、転写がRNAを鋳型とした相同組換えによる二本鎖切断修復と共役しているようである。この修復過程では、RNAポリメラーゼIIによって活発に転写されている遺伝子内の二本鎖切断が効率的かつ正確に再結合される。